Benutzer-Werkzeuge
Seitenleiste
Inhaltsverzeichnis
„Dem Gehirn zuhören“
Das Team
Anton
Severin
Tsendy (seit Dezember 2015 ausgestiegen)
Simone
Projektidee
Wir wollen ein Programm schreiben, welches EEG-Daten einliest und diese mit Hilfe von MIDI-Daten in Klänge umwandelt, dabei die verschiedensten Bereiche des Gehirns selektiv hörbar und miteinander vergleichbar macht.
Unser Ziel ist es das Programm in Echtzeit zu testen. Hierzu bietet es sich an, dem Probanden verschiedene Bilder zu zeigen, Rechenaufgaben zu stellen oder einfach ein simples Gespräch zu führen. Paralell dazu wird der Klang, welcher dem Gehirn entlockt wird aufgenommen, womöglich zeitgleich abgespielt. Hoffentlich lassen sich in den Klängen Rückschlüsse auf die Vorgänge im Kopf des Versuchskaninchens ziehen.
Hilfsmittel
Computer
EEG-Instrumente (genaueres folgt)
Programmiertechnische Hilfmittel
Datenpool von früheren EEG Messungen
MIDI Protokolle
Mathematische Hilfsmittel
Fouriermethode aus der Analysis
Nützliche Links
Arbeitsphase
19.11.2015: erste Koordinierung
- Auswertung erster Quellen (siehe Cloud)
- Grafische Darstellung von „Messdaten aus der Dose“ durch python 2.7
- Festlegung eines ersten Zeitplans:
Dienstag 24.11.2015 13 Uhr Treffen mit Stefan Born Crashkurs zu Fourier (Analysis)
Langfristiges Ziel:
Bis Februar eine funktionstüchtige Software programmiert zu haben.
Simone kümmert sich um ein Briefing durch befreundete Neurologin.
Erste Gedanken zur Realisierung:
Differenzierung der verschiedenen Aufgabenbereiche (Sinneswahrnehmung, Unterbewusstsein, Emotionen etc.) des Gehirns, die entsprechenden Elektroden gruppieren und verschiedenen Parametern des MIDI-Protokolls zuordnen. Beispiel: Frequenz / Amplitude aus Sinneswahrnehmung umwandeln in Tonhöhe/Ambitus; Amplitude aus Emotionen umwandeln in Lautstärke des Tones; Frequenz des „Emotionenzentrums“ in Freuqenz eines Oszillartors.
Zusammenrechnung aller outputs zu einer Masterkurve?
Probleme:
- Einlesen der Messdaten simultan, ohne Latenz
- Zuweisung der unterschiendlichen Parametern
- Konvertierung in ein MIDI file; weiterhin ohne Latenz
- Einspeisung der MIDI files in einen Softwaresynthesizer, in der Hoffnung etwas mehr als weißes Rauschen zu hoeren…
26.11.2015: Los geht's!
Nachdem Stefan uns am vergangenen Dienstag einen kurzen Überblick über die Fourier-Transformation geliefert hat; verfügen wir nun über den nötigen Überblick und konnten mit der Aufteilung der Aufgaben beginnen.
Hierbei legten wir folgende, vorläufige Etappenziele fest:
- Input→ Dateien simultan einlesen; in geeignetes Format bringen, Weiterleitung (import)
- Fourier Analyse; Rekonstruktion der Funktionsstücke
- event. grafische Darstellung des Frequenzspektrums
- Umwandlung/Konvertierung in MIDI bzw. Weitergabe an Softwaresynthesizer
Probleme des Tages:
- Auslesung der offiziellen EEG-Daten in Python nicht ohne weiteres möglich → Festlegung auf ein „Standartformat“ erforderlich
- Verstehen der Fourier-Analysis
- Zweifel an der Sinnhaftigkeit einer Übertragung ins MIDI-Protokoll #gefixt
03.12.2015:Ansätze gesucht...
Da es in letzter Zeit immer häufiger zu Problemen mit .EEG - Formaten kam, haben wir uns heute intensiver mit den verschiedenen Datentypen und deren Inhalte beschäftigt. Die Dateien des EEG-Herstellers Neurowerk noch zu große Probleme bei der Auswertung verursachen, haben wir beschlossen erst einmal mit den Daten von Stefan zu arbeiten (siehe Owncloudverzeichnis Unterordner eeg).
Dateien einlesen
Die ersten Probleme entstanden bereits beim Einlesen der Datei. Severin hat sich hierbei um die Entschlüsselung der .mat Datein gekümmert. Um Matlab-Dateien in Python einzulesen genügt schon folgender Codeschnippsel:
import scipy.io\\ mat = scipy.io.loadmat('DATEI.mat')
entsprechend erhält man als Output:
{'cnt': array([[ -40, -15, -18, ..., -20, -20, -18], [-339, -119, -152, ..., -168, -177, -158], [-822, -301, -345, ..., -378, -406, -369], ..., [1505, 1356, 987, ..., 415, -2, 300], [1566, 1445, 990, ..., 468, 15, 379], [1510, 1428, 878, ..., 375, -125, 295]], dtype=int16), 'nfo': array([[ ([[100]], [[array([u'left'], dtype='<U4'), array([u'foot'], dtype='<U4')]], [[array([u'AF3'], dtype='<U3'), array([u'AF4'], dtype='<U3'), array([u'F5'], dtype='<U2'), array([u'F3'], dtype='<U2'), array([u'F1'], dtype='<U2'), array([u'Fz'], dtype='<U2'), array([u'F2'], dtype='<U2'), array([u'F4'], dtype='<U2'), array([u'F6'], dtype='<U2'), array([u'FC5'], dtype='<U3'), array([u'FC3'], dtype='<U3'), array([u'FC1'], dtype='<U3'), array([u'FCz'], dtype='<U3'), array([u'FC2'], dtype='<U3'), array([u'FC4'], dtype='<U3'), array([u'FC6'], dtype='<U3'), array([u'CFC7'], dtype='<U4'), array([u'CFC5'], dtype='<U4'), array([u'CFC3'], dtype='<U4'), array([u'CFC1'], dtype='<U4'), array([u'CFC2'], dtype='<U4'), array([u'CFC4'], dtype='<U4'), array([u'CFC6'], dtype='<U4'), array([u'CFC8'], dtype='<U4'), array([u'T7'], dtype='<U2'), array([u'C5'], dtype='<U2'), array([u'C3'], dtype='<U2'), array([u'C1'], dtype='<U2'), array([u'Cz'], dtype='<U2'), array([u'C2'], dtype='<U2'), array([u'C4'], dtype='<U2'), array([u'C6'], dtype='<U2'), array([u'T8'], dtype='<U2'), array([u'CCP7'], dtype='<U4'), array([u'CCP5'], dtype='<U4'), array([u'CCP3'], dtype='<U4'), array([u'CCP1'], dtype='<U4'), array([u'CCP2'], dtype='<U4'), array([u'CCP4'], dtype='<U4'), array([u'CCP6'], dtype='<U4'), array([u'CCP8'], dtype='<U4'), array([u'CP5'], dtype='<U3'), array([u'CP3'], dtype='<U3'), array([u'CP1'], dtype='<U3'), array([u'CPz'], dtype='<U3'), array([u'CP2'], dtype='<U3'), array([u'CP4'], dtype='<U3'), array([u'CP6'], dtype='<U3'), array([u'P5'], dtype='<U2'), array([u'P3'], dtype='<U2'), array([u'P1'], dtype='<U2'), array([u'Pz'], dtype='<U2'), array([u'P2'], dtype='<U2'), array([u'P4'], dtype='<U2'), array([u'P6'], dtype='<U2'), array([u'PO1'], dtype='<U3'), array([u'PO2'], dtype='<U3'), array([u'O1'], dtype='<U2'), array([u'O2'], dtype='<U2')]], [[-0.20109028464758363], [0.20109028464758363], [-0.48547489245766257], [-0.32894736842105265], [-0.16535230938896844], [0.0], [0.16535230938896844], [0.32894736842105265], [0.48547489245766257], [-0.605915412132058], [-0.39919578789655963], [-0.1976593484504406], [0.0], [0.1976593484504406], [0.39919578789655963], [0.605915412132058], [-0.7483468341419532], [-0.5247297565962373], [-0.30963911235797154], [-0.10226302831631562], [0.10226302831631562], [0.30963911235797154], [0.5247297565962373], [0.7483468341419532], [-0.8771929824561404], [-0.6456905834155688], [-0.4215490013382115], [-0.20773756505037197], [0.0], [0.20773756505037197], [0.4215490013382115], [0.6456905834155688], [0.8771929824561404], [-0.7483468341419532], [-0.5247297565962373], [-0.30963911235797154], [-0.10226302831631562], [0.10226302831631562], [0.30963911235797154], [0.5247297565962373], [0.7483468341419532], [-0.605915412132058], [-0.39919578789655963], [-0.1976593484504406], [0.0], [0.1976593484504406], [0.39919578789655963], [0.605915412132058], [-0.48547489245766257], [-0.32894736842105265], [-0.16535230938896844], [0.0], [0.16535230938896844], [0.32894736842105265], [0.48547489245766257], [-0.10395864942009443], [0.10395864942009443], [-0.17113186141765643], [0.17113186141765643]], [[0.6865651771052461], [0.6865651771052461], [0.5254742370339627], [0.4652018297279918], [0.43208640720881303], [0.4215490013382115], [0.43208640720881303], [0.4652018297279918], [0.5254742370339627], [0.2716570424217247], [0.23384347850759124], [0.21394493707763337], [0.20773756505037197], [0.21394493707763337], [0.23384347850759124], [0.2716570424217247], [0.15177169088329834], [0.1255310315311084], [0.11086095908649396], [0.1042664794829223], [0.1042664794829223], [0.11086095908649396], [0.1255310315311084], [0.15177169088329834], [0.0], [0.0], [0.0], [0.0], [0.0], [0.0], [0.0], [0.0], [0.0], [-0.15177169088329834], [-0.1255310315311084], [-0.11086095908649396], [-0.1042664794829223], [-0.1042664794829223], [-0.11086095908649396], [-0.1255310315311084], [-0.15177169088329834], [-0.2716570424217247], [-0.23384347850759124], [-0.21394493707763337], [-0.20773756505037197], [-0.21394493707763337], [-0.23384347850759124], [-0.2716570424217247], [-0.5254742370339627], [-0.4652018297279918], [-0.43208640720881303], [-0.4215490013382115], [-0.43208640720881303], [-0.4652018297279918], [-0.5254742370339627], [-0.6558381161286909], [-0.6558381161286909], [-0.8603379652659916], [-0.8603379652659916]])]], dtype=[('fs', 'O'), ('classes', 'O'), ('clab', 'O'), ('xpos', 'O'), ('ypos', 'O')]), '__header__': 'MATLAB 5.0 MAT-file, Platform: GLNXA64, Created on: Mon Jul 7 18:38:04 2008', '__globals__': [], 'mrk': array([[ ([[2091, 2891, 3691, 4491, 5291, 6091, 6891, 7691, 8491, 9291, 10091, 10891, 11691, 12491, 13291, 16292, 17092, 17892, 18692, 19492, 20292, 21092, 21892, 22692, 23492, 24293, 25093, 25893, 26693, 27493, 30493, 31293, 32093, 32893, 33693, 34493, 35293, 36093, 36893, 37693, 38493, 39293, 40093, 40893, 41694, 44694, 45494, 46294, 47094, 47894, 48694, 49495, 50295, 51095, 51895, 52695, 53495, 54295, 55095, 55895, 58895, 59695, 60495, 61295, 62095, 62895, 63695, 64495, 65295, 66095, 66895, 67695, 68495, 69295, 70095, 73094, 73894, 74694, 75494, 76294, 77095, 77895, 78695, 79495, 80295, 81095, 81895, 82695, 83495, 84295, 87295, 88095, 88895, 89695, 90495, 91295, 92095, 92895, 93695, 94495, 97398, 98198, 98998, 99798, 100598, 101398, 102198, 102998, 103798, 104598, 105398, 106198, 106998, 107798, 108599, 111598, 112398, 113198, 113998, 114798, 115598, 116398, 117198, 117998, 118798, 119598, 120398, 121198, 121998, 122798, 125797, 126597, 127397, 128197, 128997, 129797, 130597, 131397, 132197, 132997, 133797, 134597, 135397, 136198, 136998, 139996, 140797, 141597, 142397, 143197, 143997, 144797, 145597, 146397, 147197, 147997, 148797, 149597, 150397, 151197, 154196, 154996, 155796, 156596, 157396, 158196, 158996, 159796, 160596, 161396, 162196, 162996, 163796, 164596, 165396, 168397, 169197, 169997, 170797, 171597, 172397, 173197, 173997, 174797, 175597, 176397, 177197, 177997, 178797, 179597, 182598, 183398, 184198, 184998, 185798, 186598, 187398, 188198, 188998, 189798]], [[-1, -1, -1, -1, -1, 1, -1, 1, -1, -1, -1, -1, -1, -1, -1, 1, 1, -1, 1, 1, 1, 1, 1, 1, 1, -1, -1, -1, -1, 1, 1, -1, -1, 1, 1, 1, -1, 1, -1, -1, 1, -1, 1, -1, -1, -1, 1, -1, 1, 1, 1, 1, 1, -1, 1, 1, -1, -1, 1, 1, -1, 1, -1, -1, 1, -1, -1, 1, 1, 1, -1, 1, 1, 1, -1, -1, 1, -1, -1, 1, 1, -1, 1, 1, 1, -1, 1, -1, -1, 1, -1, -1, 1, 1, 1, 1, -1, -1, -1, 1, 1, -1, -1, 1, 1, 1, 1, 1, 1, -1, 1, -1, 1, -1, 1, -1, 1, -1, 1, -1, -1, 1, -1, -1, -1, -1, -1, -1, -1, -1, -1, 1, -1, -1, -1, -1, -1, 1, -1, 1, 1, 1, -1, -1, 1, -1, 1, 1, 1, 1, 1, 1, -1, -1, 1, 1, 1, 1, -1, -1, 1, -1, -1, 1, -1, 1, -1, 1, -1, 1, -1, -1, -1, 1, -1, 1, -1, 1, -1, 1, -1, 1, -1, 1, 1, -1, -1, 1, 1, -1, -1, 1, 1, 1, 1, -1, 1, 1, -1, 1]])]], dtype=[('pos', 'O'), ('y', 'O')]), '__version__': '1.0'}
Analyse der Metadaten
Dieser Code lässt sich in verschiedene Teile unterteilen. Von Bedeutung für unser Projekt ist hier bei das 'cnt'-Array, welches die Messwerte der 59 Elektroden beeinhaltet.
Durch…
plt.plot(mat['cnt'],'3')
bzw.
plt.plot(mat['cnt'][:,0],'3')
…lassen sich die verschiedenen Ströme darstellen, zuerst die gesamte Messung aller 59 Elektroden, letzteres bezieht sich nur auf die erste Elektrode.
Neben dem numpy.array befindet sich auch ein 'nfo'(info)-Blog in der.mat Datei.
Hierbei handelt es sich um ein Array im Array im Array im Array.
In diesen etwas verrückten Unterverzeichnis werden die allgemeinen Messinformationen gespeichert, wie die Samplingfrequenz, die Position bzw. Name der Elektroden u.ä..\
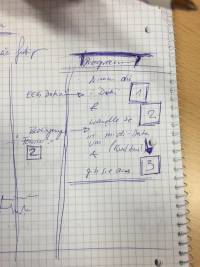
Den etwas verwirrenden Zugriff auf diese Arrays haben wir hier als Foto eingefügt:

Probleme des Tages
Dank der unnötigen Verschachtelung der 'nfo'-Typen dauerte es seine Zeit, bis wir den deren Inhalt überhaupt finden konnten. Nach einigen Probieren und suchen sowie nach der Hilfe von Arik und Stefan konnte dies schließlich auch gelingen.
10.12.2015: Erste Fortschritte - auch mit Schummeln...
Nach einer kleinen Dienstags-Einheit hat Severin es geschafft, einige Daten aus dem Datenpool in ein MIDIfile zu übersetzen.
Hierbei hat er die 59-Spaltige Matrix auf eine 4-Spalten-Matrix reduziert, die Parameter 'passend' gerechnet und als eine Liste an einen Midi-Converter (MIDITime) weitergegeben.
Die entsprechende MIDI-Dateien finden sich in unserer ownCloud, demnächst wird noch eine Audiodatei nachgereicht (siehe unten).
Somit wurde der Beweis erbracht, dass eine Umwandlung von EEG-Daten nach MIDI grundsätzlich möglich ist; sinnvolle und nachvollziehbare Signale sind dies jedoch noch nicht, das wird nach einer umfangreichen Analyse und Interpretation durch SFT/FFT hoffentlich möglich sein.
Nach einigen Spielereien hat Simone sogar eine Version der MIDI-Datei erstellt, die den Interpreter nicht überfordert und ein akzeptables bzw. hörbares Ergebnis liefert:
Wir beginnen, verschiedene Schnittstellen und Module zu programmieren:
- Input-Schnittstelle mit Weiche welche zwischen EEG-File und EEG-Gerät differenziert, Daten als np.array weitergibt
- Fourrier-Modul errechnet aus dem Array die Frequenzen aller Elektroden in Abhängigkeit zu der Zeit
- Filter-Modul (mglw. auch vor der FFT!) beeinflusst überflüssige Frequenzen, grenzt Spektrum ein
- Interpreter - Modul münzt die Frequenzen nach vorgegebenen Regeln in MIDI-Parameter um
07.01.2016: Präsentation und Bestandsaufnahme
Der heutige Mathesis-Blog wurde überwiegend genutzt um allen anderen Gruppen den Stand des eigenen Projektes näher zu bringen. Außerdem hat Anton weiter das Fourier-Modul verfeinert und konnte bereits das Input-Modul einbinden. In letzterem arbeitete Severin heute an einer Dateiformat-Weiche, durch welche das Einlesen verschiedener Datentypen möglich gemacht wird, die weitergegebenen Daten allerdings in eine einheitliche Struktur gebracht werden.
14.01.2016: In großen Schritten voraus
- erste Versuche zwei Module miteinander zu verknüpfen:
[IMPORT] ⇒ [PICKLE] ⇒ [FOURIÉR] ⇒ [PLOT] ✔
- Verbesserung des Fouriér-Moduls durch Anton, sehr funktional, strapaziert allerdings die Rechen- und Arbeitsspeicherkapazität zu sehr.
- Auswertung der Lektüre (Guide to Brain-Computer Music Interfacing)
- Aus gegebenen Anlass verschieben wir die Erstellung des Filter-Moduls, es wird nach Fertigstellung der restichen Module eingefügt bzw. produziert, um sich komplett auf die Berrechnung der Filter konzentrieren zu können.
21.01.2016: personelle Probleme und Fortschritte
- Da für diese Woche sowieso ein Treffen am Fr, 22.01. vorgesehen hatten und sich Simone und Severin abgemeldet hatten, gab es (bisher) keine enormen Fortschritte, langsam aber sicher ist die Effizienz von der Datenverarbeitung das drängendste Problem:
- HDF5-module als Alternative zu Pickle speziell für die großen Datenmengen der EEG-Daten, Implementation folgt (noch nicht in der Cloud) - die Daten müssen in kleineren Stücken zur Verarbeitung geleitet werden, auch um die massive Arbeitsspeicherauslastung zu reduzieren
- das jetzige Fourier-Programm liefert anscheinend keine nach der Zeit geordnete Ergebnisse, nur über die ganze eingelesene Datei gemittelte Häufigkeit von verschiedenen Frequenzen bzw. zeitlich sortierte Frequenzwerte. Codeschnipsel mit Kommentaren:
fourier = np.fft.fft(MatrixToArray(eegmatrix)) # 'fftfreq' gibt einen Array aus den einzelnen Frequenzen geteilt durch die Samplerate freqarray = np.fft.fftfreq(MatrixToArray(eegmatrix).size) freq = [x * samplerate for x in freqarray] '''an dieser Stelle sollte das fertige array erscheinen, allerdings scheint der Code hier die Einträge chornologisch zu sortieren'''
- Erste Ansätze zur Analyse der Fourier-Frequenzen, Ziel: Liste/Array, die pro Zeiteinheit die stärksten Frequenzen enthält näheres siehe unten!
22.01.2016: Nachtrag, theoretische Grundlagen der Interpretation von Fouriér-Datensätzen
Nach intensivsten Diskussionen lässt sich der weitere Projektverlauf durch folgende Punkte skizzieren:
- Umbau der Softwarestruktur von Pickle zu HDF5 in die Wege geleitet
- Planung einer Benutzeroberfläche, welche folgenden Voraussetzungen genügen muss:
- grafische Darstellungen aller verfügbaren Elektroden
- Maske zur Ansteuerung einer individuellen Auswahl an auszuwertenden Elektroden
- Zuweisung der Elektroden zu Prioritäten: einhergehend mit Lautstärke, während des laufenden Programms durch Schieberegler beeinflussbar
- später Möglichkeit von „Zeitrafferaufnahmen“ welche entsprechende Daten zusammenfassend wiedergeben
- komplexe Interpretation des laufenden Datenstroms aus Importmodul:
- Differenzierung der verschiedenen Schwingungstypen (α,β,γ,Δ -Wellen) folglich entsprechend unterschiedliche Zuweisung von Parametern
- (vorerst) Reduzierung aller Framerates auf 100 bzw. 10 fps, da eine rhtythmische Differenzierung so möglich gemacht wird. Fraglich bleibt, ob der Verlust der Genauigkeit zu gravierenden Einbußen führt; allerdings wiegt die reduzierte Arbeitsspeicherauslastung hierbei höher.
- Verarbeitung eines Frames von einer Elektrode:
- Fourié-Trafo liefert eine/drei Frequez(en), welche in diesem Frame hauptsächlich enthalten sind
- Schlüsselfrequenz läuft durch einen Analyzer, welcher ermittelt um welchen Schwingungstyp es sich handelt
- es folgt die Vergabe und Beeinflussung der verschiedenen Parameter durch die Frequenz
- Wie diese erfolgt gilt es noch zu klären (Gedanke: „ für xyz-Frequenz tue dies…“)
28.01.2016: Rückrudern, Benutzeroberfläche, Bugfixes
Erste Versuche, die Gedanken des letzten Treffens umzusetzen hapern teilweise noch in der Umsetzung: Import-Modul muss bedingt durch die neue Datenstruktur teilweise neu aufgesetzt werden. Es wird eine frames-weise Import-Funktion eingeführt, die es erlaubt Ressourcen zu sparen und interessante Frames zu separieren. Leider gibt es hierbei Probleme mit den .mat Formaten, da die derzeit .scipy-basierte Importfunktion eine 'scheibchenweise' Importierung nich unterstützt.
Festlegung der Benutzeroberfläche auf qt-Basis, für komplexe Zusammenhänge und Widgets wie Schieberegler und Buttons.
04.02.2016: "pricipal component analysis", GUI, HDF5
Mit Blick auf das sinnvolle Interpretieren der Fourier-Daten wendet sich Severin der PCA zu, einem Verfahren zur Strukturierung von großen Datensätzen. Somit erreichen wir auf Basis der größtmöglichen Varianz zwischen den Fourier-Frequenzen, welche als Vektoren geführt werden, gute Möglichkeiten um hörenswerte Spitzenwerte zu isolieren.
Simone befasst sich unterdessen weiter mit der Benutzeroberfläche. Der Schwerpunkt liegt hierbei auf dem individuellen Auswählen der verschiedenen Elektroden durch eine interaktive Oberfläche, sowie die simultane Beeinflussung der unterschiedlichen Prioritäten. Dies stellt sich allerdings als extrem aufwändig heraus, sofern man für alle 59 Elektroden genannte Funktionen bereitstellen möchte. Daher ist die Frage zu klären, wie mit diesem Problem umgegangen wird.
Anton ist unterdessen mit der Errichtung der Datenbankstruktur beschäftigt. Nähere Details hierbei werden folgen.
11.02.2016: spontane Fourié-Rettung
Leider kam es zu einigen terminlichen Komplikationen, sodass nur Severin kurz zum Laborblock kommen konnte. In dieser Zeit gelang es ihm gemeinsam mit Stefan das Problem der sortierten Fourier-Listen zu beheben. Hierbei stiegen wir von der ursprünglich numpy-basierter FastFourierTransformation um auf folgende, von scipy gestellter Funktion:
from scipy.signal import spectrogram freq,_,fourier = spectrogram(eegmatrix[0,:],nperseg=64, fs=samplerate)
Entsprechender Code liefert nun die Ergebnisse der Fourié - Trafo in einer Matrix und lässt zusätzlich die Möglichkeit offen, diese scheibchenweise weiterzugeben.